Optimiser les outils de simulations pour mieux évaluer les dommages radio-induits à l’ADN
Parmi les causes des effets induits par les rayonnements ionisants sur les organismes vivants, les dommages à l'ADN sont considérés comme étant à l'origine de nombreuses conséquences biologiques. Bien que très efficaces dans la prédiction de ces effets, les techniques de simulations numériques actuelles exigent un temps de calcul trop important pour prédire les dommages sur l'ADN à large échelle. C'est pour résoudre cette problématique que le Laboratoire de dosimétrie des rayonnements ionisants (LDRI) de l'IRSN a développé une nouvelle approche de simulation et l'a adaptée à son code de calcul. Cette étude est parue dans la revue Medical Physics en 2020.
Cellules irradiées au microfaisceau MIRCOM de Cadarache. Source : IRSN.
Les dommages radio-induits à l’ADN sont issus soit d’un effet direct, c’est à dire d’un dépôt d’énergie issu de l’interaction rayonnement matière dans les constituants moléculaires de l’ADN ; soit d’un effet indirect, c’est-à-dire de la réaction chimique d'espèces radiolytiques issues des ionisations et excitations au voisinage de la cible biologique avec les constituants de l’ADN. Les dommages indirects contribuent de manière très significative à l’ensemble des dommages à l’ADN avec 60 à 90% des cassures de brin produites par cette voie.
La simulation numérique, une méthode puissante mais lente
Parmi les techniques développées pour mesurer les dommages à l’ADN, la simulation numérique constitue une méthode puissante pour la prédiction de ces dommages selon le type de rayonnement (particule, énergie, fluence). De nombreux codes, basés souvent sur la méthode Monte Carlo (MC), sont développés pour réaliser ces modélisations. Geant4-DNA est une extension du code généraliste MC Geant4 ayant pour objectif la mise en « open source » des outils nécessaires à cette simulation. En particulier, l’exemple appelé dnadamage1 développé par l’IRSN et téléchargeable avec Geant4, montre comment réaliser la simulation de l’étape physique (effet direct) ainsi que l’étape chimique (effet indirect) sur une cible d’ADN représentant une portion de fibre de chromatine formée par 18 nucléosomes placés de manière hélicoïdale pour un total de 3640 paires de nucléotides et une longueur de 40 nm.
Pour la simulation de l’étape physique, Geant4-DNA permet le choix des sections efficaces des particules chargées grâce à des options de physique. Dans dnadamage1, un seuil en énergie déposée dans le volume du squelette du nucléotide est fixé afin de comptabiliser le nombre de cassures créées par effet direct.
Lors de l’étape chimique, il est considéré que seule une fraction des réactions entre les radicaux hydroxyl et le sucre de l’ADN peut induire un dommage. Dans cette étape, la seule option disponible utilise la méthode connue sous le nom de SBS (Step by step) dans laquelle, une fois les différentes espèces radiolytiques créées, la simulation de leur diffusion dans le milieu continu d’eau liquide (en utilisant les équations de Smoluchowski) est combinée à un calcul de probabilité de réaction chimique de la forme « totalement contrôlée par la diffusion » entre espèces radiolytiques présentes ou avec les constituants de l’ADN considérés statiques (figure 1).
La simulation est discrétisée en pas de temps pour limiter la diffusion libre des radicaux et évaluer les possibilités de réaction à partir de la distance qui sépare chaque radical des autres molécules. Ces pas en temps peuvent être choisis en fonction du temps global. Ainsi, au début de l’étape chimique ils sont recommandés très courts (0,35 ps pour les temps inférieurs à 1 ps) de manière à ne pas « rater » des réactions entre des espèces très proches lors de leur création, alors qu’ils peuvent être augmentés au fur et à mesure que les radicaux s’éloignent de leur lieu d’origine (10 ps pour les temps supérieurs à 10 ns).
Cette méthode de simulation très précise a été validée notamment par des comparaisons avec les valeurs des rendements radiolytiques de chaque espèce obtenus expérimentalement à différents temps après irradiation. Malheureusement elle est extrêmement gourmande en temps de calcul ce qui rend très difficile d’étendre la simulation à l’ensemble d’une population cellulaire, par exemple.
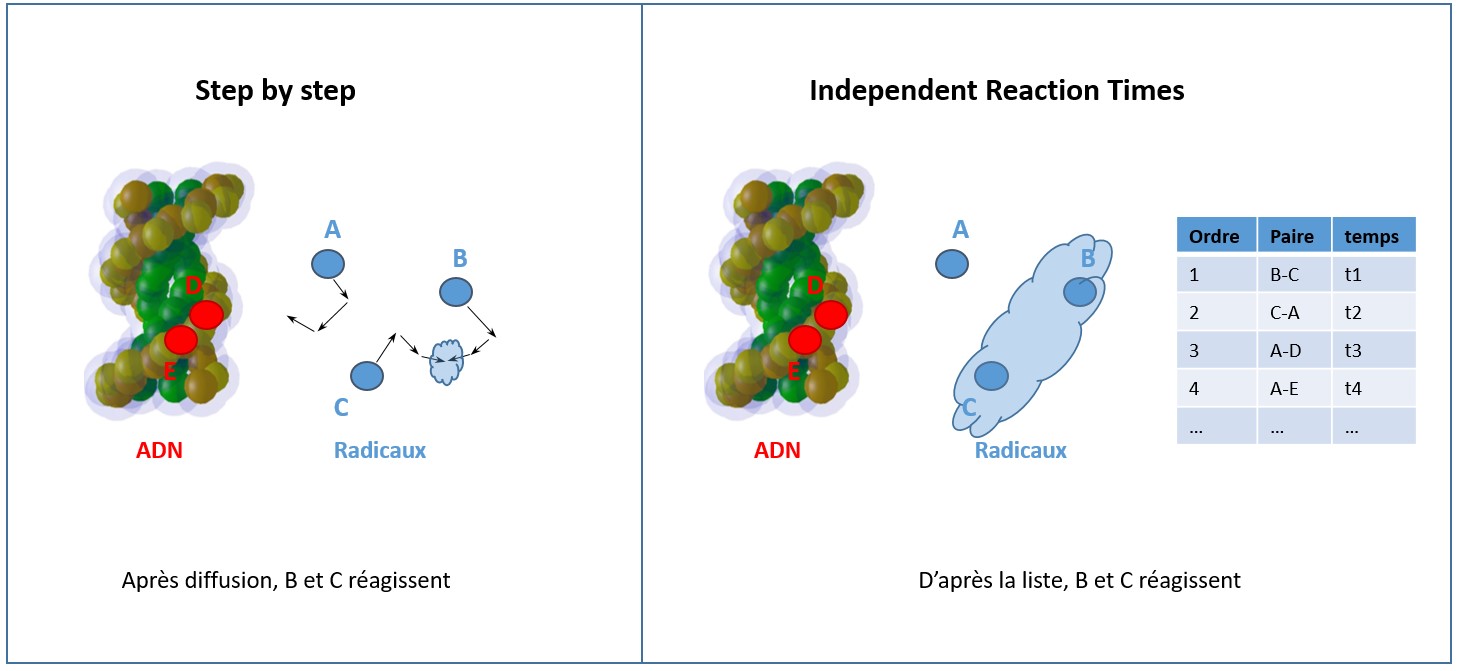
Figure 1 : Schéma du principe des techniques SBS et IRT
Optimiser le temps de calcul en ordonnant les réactifs
Le travail présenté dans l’étude publiée dans la revue Medical Physics montre l’adaptation dans Geant4-DNA d’une autre méthode de simulation de l’étape chimique appelée IRT (Independent Reaction Times). L’IRT ne simule pas la diffusion des espèces radicalaires de manière explicite. A partir de chaque position initiale, un temps de réaction est calculé pour chaque paire de réactifs. Les différentes réactions (qui peuvent être totalement contrôlées par diffusion ou partiellement, élargissant les possibilités de la simulation) sont alors ordonnées puis réalisées chronologiquement (figure 1), ce qui réduit de manière considérable le temps de calcul qui est principalement dépensé dans la mise à jour de la liste de réactions.
Cette méthode repose sur l’hypothèse que chaque réaction entre deux réactants est indépendante du système, elle n’est donc exacte que pour un système à deux réactants. Des adaptations ont donc été nécessaires afin de prendre en compte la compétition entre les différentes réactions au fur et à mesure que les distances entre les espèces varient.
Les résultats obtenus en termes de cassures de brin sur la portion de fibre de chromatine de l’exemple dnadamage1 par cette nouvelle méthode IRT ont été comparés aux résultats obtenus avec la méthode SBS actuelle de Geant4. Comme il est montré sur la figure 2, ces résultats sont très proches pour des temps de simulation de l’étape chimique de 1 ps à 3 nanosecondes. A partir de ce temps, les différences sont de l’ordre de 10% ce qui reste, néanmoins, une différence inférieure à l’incertitude associée à la détection des cassures par les techniques expérimentales. Il faut également indiquer, qu’afin de prendre en compte de manière approximée les effets d’élimination des radicaux dans le milieu cellulaire, différent chimiquement de l’eau liquide pure, la simulation de l’étape chimique est actuellement arrêtée à un temps global de 2,5 ns pour lequel les différences entre la méthode SBS et l’IRT présentée dans ce papier sont acceptables. Au vu de ces résultats, cette méthode IRT a été intégrée aux outils de simulation de l’IRSN pour gagner en temps de calcul et fera partie du prochain release de Geant4 (v11.0).
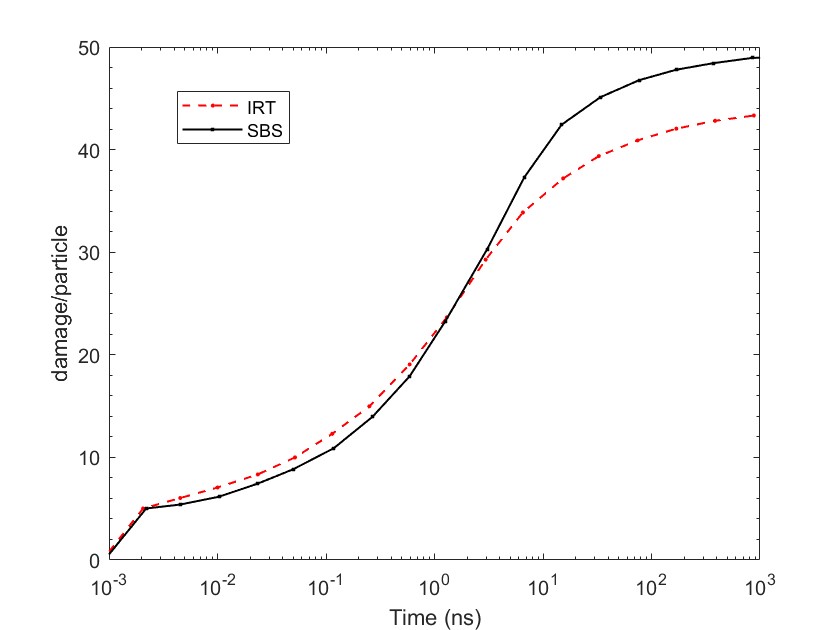
Figure 2 : Comparaison du nombre de dommages à l’ADN en fonction du temps de l’étape chimique incluant les dommages indirects calculés avec la méthode actuelle (SBS) et la nouvelle méthode présentée dans ce papier (IRT). Pour les temps typiques de simulation de l’étape chimique (2,5 ns), les deux méthodes donnent des résultats comparables.
Pour aller plus loin :
- Retrouver l’article “Assessment of DNA damage with an adapted independent reaction time approach implemented in Geant4‐DNA for the simulation of diffusion‐controlled reactions between radio‐induced reactive species and a chromatin fiber” sur le site de l’éditeur
- Découvrir les activités du LDRI
